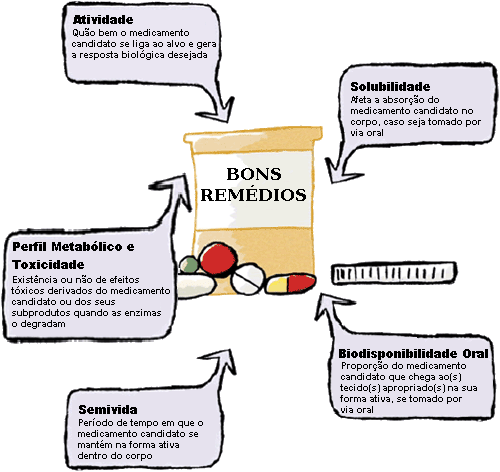
As Estruturas da Vida
 As Estruturas da Vida em PDF
As Estruturas da Vida em PDFCapítulo 4: Design de medicamentos baseado na estrutura: Do computador ao hospital
Em 1981, os médicos reconheceram uma nova e estranha doença nos EUA. Os primeiros pacientes sofriam de tipos raros de cancro e pneumonia. À medida que a doença se espalhou, os cientistas descobriram a sua causa: um vírus que ataca as células imunitárias humanas. Hoje uma das principais causas de morte a nível global, a doença é mais conhecida pelo seu acrónimo, SIDA.
O SIDA, ou Síndrome da Imunodeficiência Adquirida, é causado pelo vírus da imunodeficiência humana, ou VIH.
Embora os investigadores não tenham encontrado uma cura para o SIDA, a Biologia Estrutural aumentou em muito a compreensão do VIH e tem tido um papel determinante no desenvolvimento de medicamentos para tratar esta doença mortal.
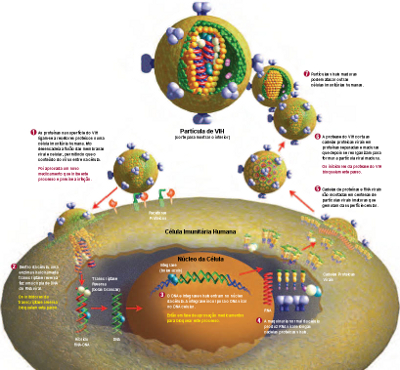
A vida de um vírus do SIDA
AIDS Vaccine Program, National Cancer Institute (Frederick, Maryland)
Clique para ampliar
O VIH foi rapidamente reconhecido como sendo um retrovírus, um tipo de vírus que transporta o seu material genético não como DNA, como a maioria dos outros organismos do planeta, mas como RNA. Depois de entrar numa célula, os retrovírus "transcrevem de forma reversa" o seu RNA para DNA.
Muito antes de se ouvir falar do VIH, já investigadores em laboratórios de todo o mundo estudavam retrovírus, alguns dos quais causadores de cancros nos animais. Estes cientistas determinaram o ciclo de vida dos retrovírus e identificaram as proteínas chave que estes vírus usam para infetar as células.
Quando o VIH foi identificado como um retrovírus, estes estudos deram uma vantagem aos investigadores do SIDA. As proteínas virais previamente identificadas tornaram-se os primeiros alvos dos medicamentos.
Revelando o alvo
Os modelos moleculares da protease do VIH neste capítulo foram gerados por Alisa Zapp Machalek.
A nossa história começa em 1989, quando os cientistas determinaram a estrutura cristalográfica de raios-X da protease do VIH, uma enzima essencial no ciclo de vida do VIH. Os cientistas farmacêuticos esperavam que, ao bloquear esta enzima, poderiam prevenir a disseminação do vírus pelo corpo.
Com a estrutura da protease do VIH ao alcance, os investigadores deixaram de trabalhar às escuras. Puderam finalmente ver a enzima alvo – com emocionantes detalhes coloridos. Ao inserir a informação estrutural num programa de modelação computacional, foi-lhes possível rodar o modelo da enzima nas três dimensões, ampliar a mesma focando átomos específicos, analisar as suas propriedades químicas e até mesmo remover ou alterar certas partes da proteína.
Mais importante ainda, podiam usar a estrutura computorizada como referência para determinar o tipo de moléculas que poderiam bloquear a enzima. Estas moléculas podem ser encontradas em bases de dados de químicos ou desenhadas num ecrã de computador e depois sintetizadas num laboratório. Estas estratégias de design de medicamentos baseadas na estrutura têm o potencial de poupar anos e milhões de dólares quando comparadas com o processo tradicional de desenvolvimento de medicamentos baseado na prática de tentativa-erro.
Estas estratégias funcionaram no caso dos inibidores da protease do VIH. "Penso que é uma notável história de êxito", diz Dale Kempf, um químico envolvido no programa de inibidores da protease do VIH nos laboratórios Abbott. "Desde a identificação da protease do VIH como alvo em 1988 até ao princípio de 1996, a colocação no mercado destes medicamentos demorou menos de 8 anos." Normalmente, no desenvolvimento de um novo medicamento passam-se entre 10 a 15 anos e gastam-se mais de 800 milhões de dólares.
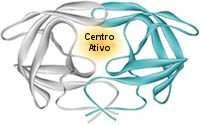
 A estrutura da protease do
VIH revelou um facto crucial – tal como as borboletas, a enzima é composta por duas metades iguais. Na maioria das
moléculas simétricas como estas, as duas metades possuem uma "área de ação", ou
centro ativo, que desempenha a função da enzima. Mas a protease do VIH possui
apenas um centro ativo – no centro da molécula, onde as duas metades se encontram.
A estrutura da protease do
VIH revelou um facto crucial – tal como as borboletas, a enzima é composta por duas metades iguais. Na maioria das
moléculas simétricas como estas, as duas metades possuem uma "área de ação", ou
centro ativo, que desempenha a função da enzima. Mas a protease do VIH possui
apenas um centro ativo – no centro da molécula, onde as duas metades se encontram.
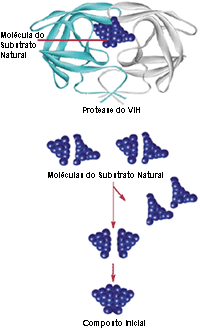
Os cientistas farmacêuticos sabiam que podiam aproveitar esta característica. Se pudessem inserir uma pequena molécula neste centro ativo, conseguiriam inativar toda a enzima – e, teoricamente, interromper a proliferação do vírus no corpo.
Várias companhias farmacêuticas começaram por usar a forma da enzima como guia. "Desenhámos moléculas candidatas a medicamento que tinham a mesma simetria bilateral da protease do VIH", diz Kempf. "Conceptualmente, pegámos no substrato natural da enzima [as moléculas sobre as quais atua], cortámos essas moléculas ao meio, girámos os fragmentos 180° e colámos as duas partes de novo."
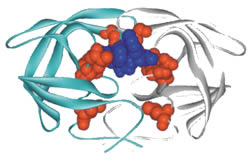
Para deleite dos investigadores, a primeira molécula que sintetizaram encaixava perfeitamente no centro ativo da enzima. Era também um excelente inibidor – prevenia o funcionamento normal da protease do VIH. Mas não era solúvel em água, o que queria dizer que não podia ser absorvida pelo corpo e não podia ser efetiva como medicamento.
Os cientistas da Abbott continuaram a modificar a estrutura desta molécula para melhorar as suas propriedades. Eventualmente conseguiram obter uma molécula não simétrica à qual deram o nome de ritonavir (Norvir®).
Uma esperança para o futuro
Entre dezembro de 1995 e março de 1996, a FDA (Food and Drug Administration) aprovou os três primeiros inibidores da protease do VIH: o Invirase™ (saquinavir) da Hoffman-La Roche, o Norvir™ (ritonavir) da Abbott e o Crixivan® (indinavir) da Merck & Co. Inicialmente, estes medicamentos foram aclamados como a primeira esperança real para os pacientes de SIDA em 15 anos. As manchetes dos jornais previam que o SIDA até poderia ter cura.
Apesar de os inibidores da protease do VIH não se terem tornado a cura milagrosa que muitos esperavam, eles representam um triunfo na terapia antiviral. Os antibióticos que são usados no tratamento de doenças causadas por bactérias são muito abundantes (apesar de estarem a ficar cada vez menos eficientes à medida que as bactérias vão desenvolvendo resistência), mas os médicos possuem muito poucos medicamentos para tratar infeções de origem vírica.
Os inibidores das proteases são também notáveis porque são um exemplo clássico de como a Biologia Estrutural pode aumentar e melhorar os métodos tradicionais de desenvolvimento de medicamentos. "Eles mostram que, com apenas algumas noções acerca da estrutura e o desenho racional de medicamentos, combinados com a química médica tradicional, se podem obter medicamentos potentes que funcionam da maneira prevista", diz Kempf.
"Isso não quer dizer que todos os problemas já estejam resolvidos", continua. "Mas claramente estes compostos tiveram um impacto profundo na sociedade". A taxa de mortalidade por SIDA diminuiu dramaticamente após a disponibilização destes medicamentos. Hoje em dia, os inibidores da protease são receitados em conjunto com outros medicamentos anti-VIH, de modo a criar um "cocktail combinado", que é mais eficiente na repressão do vírus do que cada medicamento individualmente.
Design de medicamentos baseado na estrutura: Bloquear a fechadura

Tradicionalmente, os cientistas identificam novos medicamentos manipulando os já existentes ou testando milhares de compostos em laboratório. Se pensarmos que a molécula alvo – a protease do VIH, neste caso – possui uma fechadura, esta abordagem assemelhar-se-á a uma tentativa de desenhar uma chave que encaixe na perfeição nessa fechadura a partir de uma enorme quantidade de pequenos pedaços de metal, cola e alicates.
Ao usar uma estratégia baseada na estrutura, os investigadores têm uma vantagem inicial. Começam com um modelo computorizado da estrutura tridimensional detalhada da fechadura e da sua chave (a molécula natural, chamada substrato, que encaixa na fechadura e desencadeia a replicação viral). Depois, os cientistas tentam desenhar uma molécula que vá bloquear a fechadura e impedir a ligação do substrato chave.
Conhecendo a forma tridimensional exata da fechadura, os cientistas podem descartar todos os restos de metal (pequenas moléculas) que não tenham o tamanho ou forma adequados a ela. Podem até desenhar uma pequena molécula que encaixe precisamente na fechadura. Tal molécula pode ser o ponto de partida para aqueles investigadores farmacêuticos que estão a desenhar medicamentos para o tratamento da infeção por VIH.

Claro que as moléculas biológicas são muito mais complexas do que chaves e fechaduras e o corpo humano pode reagir de forma imprevisível a novos medicamentos. Portanto, o caminho desde o ecrã do computador até à prateleira da farmácia é longo e acidentado.
Cercando a resistência
O VIH é um alvo em movimento. Quando se reproduz dentro de um organismo, em vez de gerar réplicas exatas de si mesmo, produz uma variedade de partículas virais ligeiramente alteradas. Alguns destes mutantes são capazes de escapar, ou "resistir", aos efeitos dos medicamentos e podem transmitir essa resistência à sua descendência. Embora a maioria das partículas virais sucumba inicialmente ao medicamento, estes mutantes resistentes sobrevivem e multiplicam-se. O medicamento eventualmente perde a sua atividade anti-VIH porque a maioria das partículas virais da pessoa infetada lhe são resistentes.
Alguns investigadores estão a trabalhar numa nova geração de inibidores da protease do VIH desenhados para combater estirpes específicas resistentes aos medicamentos.
Imagens computorizadas detalhadas da protease do VIH destas estirpes, revelaram que mesmo algumas substituições de aminoácidos longe do centro ativo da enzima podem gerar resistência. Alguns grupos de investigação estão a tentar combater a enzima no seu próprio jogo, desenhando medicamentos que se liguem a estas formas mutantes de protease do VIH. Outros estão a tentar desenhar moléculas que se liguem ao calcanhar de Aquiles da molécula – os ácidos aspárticos do centro ativo e outros aminoácidos que, se alterados, inutilizam a enzima. Outros ainda, estão a tentar descobrir inibidores que sejam mais potentes e convenientes de tomar, que tenham menos efeitos secundários ou que combatam melhor as estirpes mutantes do vírus.
Controlar a dor da artrite
Embora os inibidores da protease do VIH sejam exemplos clássicos do design de medicamentos baseado na estrutura, são também pouco comuns (pelo menos por enquanto. Apesar de muitas companhias farmacêuticas possuírem divisões inteiras devotadas à Biologia Estrutural, a maioria recorre a uma abordagem complementar, aliada a outros métodos mais tradicionais de descoberta de medicamentos. Em muitos casos, a estrutura de uma molécula alvo é determinada depois de uma triagem tradicional ou mesmo depois do medicamento estar no mercado.

National Institutes of Health
Este foi o caso do Celebrex®. Inicialmente desenvolvido para o tratamento da artrose e da artrite reumatoide adulta, o Celebrex® tornou-se o primeiro medicamento a ser aprovado para o tratamento de uma doença chamada FAP ou polipose adenomatosa familiar, que leva a cancro do cólon.
Normalmente, a dor e inchaço da artrite são tratados com medicamentos como a aspirina ou o ibuprofeno, os chamados anti-inflamatórios não esteroides (AINEs). Mas este tipo de medicação pode danificar o sistema gastrointestinal, incluindo úlceras hemorrágicas. De facto, num estudo recente descobriu-se que esses efeitos secundários levam a mais de 100 000 hospitalizações e 16 500 mortes anuais. De acordo com este estudo, se estes efeitos secundários fossem incluídos nas tabelas de mortalidade, seriam a 15ª causa de morte nos EUA.
Uma descoberta afortunada permitiu aos cientistas desenvolverem medicamentos que retêm as propriedades anti-inflamatórias dos AINEs, sem os efeitos secundários causadores de úlceras.
Ao estudar estes medicamentos a nível molecular, os AINEs bloqueiam a ação de duas enzimas relacionadas designadas ciclo-oxigenases. A designação abreviada destas enzimas é COX-1 e COX-2.
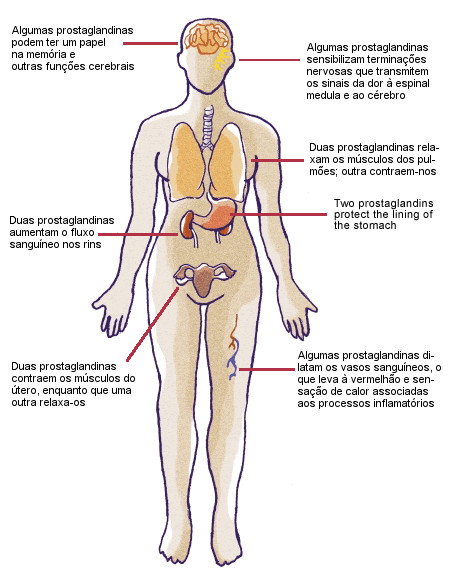
Embora estas enzimas partilhem algumas funções, também diferem em alguns aspetos importantes. A COX-2 é produzida em resposta a uma lesão ou infeção e ativa moléculas que iniciam as respostas inflamatórias e imunitárias. Ao bloquear a COX-2, os AINEs reduzem a inflamação e a dor causadas pela artrite, dores de cabeça e entorses.
Em contraste, a COX-1 produz moléculas, designadas por prostaglandinas, que protegem o revestimento estomacal dos ácidos digestivos. Quando os AINEs bloqueiam esta função, provocam a formação de úlceras.
Para poderem criar um analgésico que não provoque o aparecimento de úlceras, os cientistas perceberam que havia a necessidade de desenvolver novos medicamentos que bloqueassem a COX-2, mas que não interferissem com a função da COX-1. Tal composto foi descoberto utilizando a química medicinal corrente, tendo sido comercializado sob o nome de Celebrex®. Rapidamente se tornou o fármaco mais vendido na história dos EUA, gerando mais receitas médicas no seu primeiro ano de mercado do que os dois medicamentos líderes nessa altura combinados.
Simultaneamente, os cientistas estavam a estudar a estrutura molecular das enzimas COX. Através da Biologia Estrutural puderam verificar exactamente como é que o Celebrex® se liga à COX-2, mas não à COX-1.
As estruturas tridimensionais da COX-2 e da COX-1 são quase idênticas. Mas, existe uma alteração num aminoácido no centro ativo da COX-2 que cria uma reentrância extra que permite ligações. É a este local que o Celebrex® se liga.
Para além de mostrar aos investigadores, com detalhe átomo a átomo, como é que o medicamento se liga ao seu alvo, as estruturas das enzimas COX vão continuar a servir de modelo para perceber como é que estas moléculas funcionam no corpo.
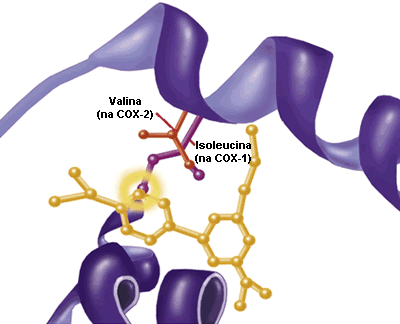
Adaptado com autorização da revista Nature ©1996 Macmillan Magazines Ltd.
Perfil de um Estudante: O fascínio pela Infeção
"Eu gosto mesmo de estudar retrovírus", diz Kristi Pullen, que se licenciou em Bioquímica pela University of Maryland, Baltimore County (UMBC). "Também gosto de agentes altamente infecciosos, como o Ébola. Quanto mais virulento, menos conhecido e, portanto, mais sujeito a todo o tipo de questões fascinantes. Não poderia deixar de me interessar."

Cortesia de Kelly Burns Photography, Columbia, Maryland
Para além do seu trabalho relacionado com as aulas na UMBC, Pullen ajudou na determinação da estrutura de retrovírus no laboratório de espetroscopia de RMN de Michael Summers. Esta investigação consiste em saber como os retrovírus fazem o empacotamento de "ogivas de RNA", que é o que lhes permite espalharem-se pelo corpo. O trabalho poderá eventualmente levar a um novo alvo para medicamentos para doenças retrovirais, incluindo o SIDA.
Estudante de Doutoramento
University of California, Berkeley
Até ao seu último ano do Ensino Secundário, Pullen queria ser uma cirurgiã de Ortopedia. Mas, após a sua primeira experiência de trabalho num laboratório, reconheceu que "existe mais em ciência do que em Medicina". Depois de ter feito algumas disciplinas de ciências, apercebeu-se de que tinha uma necessidade interior de aprender ciência e trabalhar num laboratório.
Pullen é agora uma estudante de Doutoramento na University of California, Berkeley no Departamento de Biologia Celular e Molecular. Planeia continuar a estudar Biologia Estrutural, terminar o Doutoramento e, quem sabe, até fazer depois Medicina.
Tem também alguns objetivos a longo prazo. "Em última análise, o que gostaria de fazer bem bem bem lá para a frente, seria liderar os NIH [National Institutes of Health, Institutos Nacionais de Saúde] ou os CDC [Centers for Disease Control and Prevention, Centros de Controlo e Prevenção de Doenças] e, dessa forma, ter influência na saúde de um grande número de pessoas – de todo o país."
O que é o design de medicamentos baseado na estrutura?
Como é que o design de medicamentos baseado na estrutura foi utilizado no desenvolvimento de um inibidor da protease do VIH?
De que modo é que a diferença estrutural entre a COX-1 e a COX-2 é responsável pela eficácia do Celebrex®?
Como é que os vírus se tornam resistentes aos medicamentos?
Tradução conjunta de Diana Barbosa e da equipa coordenada por José Pissarra (Maria Susana Jorge Pereira, Luís Gustavo de Carvalho Pereira,
Maria Fernanda Fidalgo Ferro de Beça, Armando Jorge Gomes Teixeira, Armando Jorge Gomes Teixeira e Fernando Manuel dos Santos Tavares).
Revisão científica de Maria João Guimarães Fonseca.